【R】stacks
1. はじめに
stacksは、機械学習におけるアンサンブルで様々なモデルを組み合わせて文字通り[stack]するパッケージです。
使い方は、紹介ページにあるように
1.rsample, parsnip, workflows, recipesや tuneの機能を使って、候補となるモデルの組み合わせを定義します。
2.stacks()関数を使ってdata_stackオブジェクトを初期化
3.data_stackオブジェクトに、add_candidate()関数でモデルをstackしていきます。
4.blend_predictionにて推測をどのように組み合わせるか評価します。
5.fit_members()関数で学習します。
6.predict()関数で推測します。
2. インストール
Githubからインストールします。
remotes::install_github("tidymodels/stacks", ref = "main")3. 使ってみる
vignettesのbasics.Rmdを参考に使ってみます。データは、アカメアマガエル(Red-eyed tree frog)の孵化の条件をデータ化したものです。目的変数は、孵化までの時間(latency)です。
3.1 データの準備
データを読み込み、不要なNAを除去します。
library(tidymodels)
library(tidyverse)
library(stacks)
library(kernlab)
data("tree_frogs")
tree_frogs <- tree_frogs %>%
filter(!is.na(latency)) %>%
select(-c(clutch, hatched))
force(tree_frogs)
> force(tree_frogs)
# A tibble: 1,212 x 7
clutch treatment reflex age t_o_d hatched latency
<fct> <chr> <fct> <dbl> <chr> <chr> <dbl>
1 168 control full 466965 morning yes 22
2 145 gentamicin full 404310 afternoon no NA
3 149 gentamicin full 426220 night no NA
4 100 control mid 355360 night no NA
5 230 gentamicin mid 356535 night no NA
6 99 control low 361180 night yes 360
7 145 gentamicin full 400070 afternoon no NA
8 133 control full 401595 afternoon yes 106
9 100 control mid 357810 night yes 180
10 182 control mid 358410 night no NA
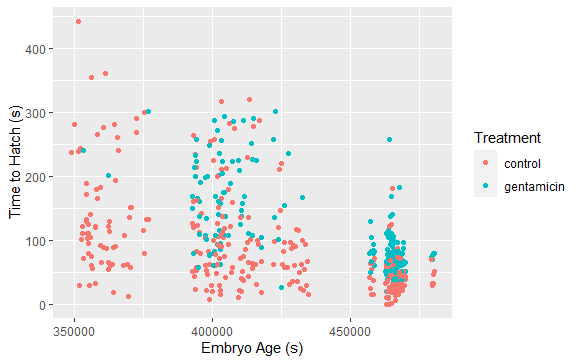
# ... with 1,202 more rows一旦、グラフに表示してどのようなデータか見てみます。
library(ggplot2) ggplot(tree_frogs) + aes(x = age, y = latency, color = treatment) + geom_point() + labs(x = "Embryo Age (s)", y = "Time to Hatch (s)", col = "Treatment")
set.seed(123) tree_frogs_split <- initial_split(tree_frogs) tree_frogs_train <- training(tree_frogs_split) tree_frogs_test <- testing(tree_frogs_split) folds <- vfold_cv(tree_frogs_train, v = 5) tree_frogs_rec <- recipe(latency ~ ., data = tree_frogs_train) %>% step_dummy(all_nominal()) %>% step_zv(all_predictors()) tree_frogs_wflow <- workflow() %>% add_recipe(tree_frogs_rec) metric <- metric_set(rmse) ctrl_grid <- control_stack_grid() ctrl_res <- control_stack_resamples()
3.2 モデル候補の定義
モデルを定義していきます。今回は、lin_reg、random forest, SVMの3つのモデルを使ってみます。
まずは、リニアモデル
lin_reg_spec <-
linear_reg() %>%
set_engine("lm")
lin_reg_wflow <-
tree_frogs_wflow %>%
add_model(lin_reg_spec)
lin_reg_res <-
fit_resamples(
lin_reg_wflow,
resamples = folds,
metrics = metric,
control = ctrl_res
)
次にRandomForestモデルです。
lin_reg_spec <-
linear_reg() %>%
set_engine("lm")
lin_reg_wflow <-
tree_frogs_wflow %>%
add_model(lin_reg_spec)
lin_reg_res <-
fit_resamples(
lin_reg_wflow,
resamples = folds,
metrics = metric,
control = ctrl_res
)
最後に、SVMです。
svm_spec <-
svm_rbf(
cost = tune(),
rbf_sigma = tune()
) %>%
set_engine("kernlab") %>%
set_mode("regression")
svm_wflow <-
tree_frogs_wflow %>%
add_model(svm_spec)
set.seed(1)
svm_res <-
tune_grid(
svm_wflow,
resamples = folds,
grid = 5,
control = ctrl_grid
)
オブジェクトの初期化。
stacks()
> stacks()
# A data stack with 0 model definitions and 0 candidate members.モデル候補を積み上げていきます。
tree_frogs_data_st <- stacks() %>% add_candidates(lin_reg_res) %>% add_candidates(rf_reg_res) %>% add_candidates(svm_res) tree_frogs_data_st
> tree_frogs_data_st
# A data stack with 3 model definitions and 7 candidate members:
# lin_reg_res: 1 sub-model
# rf_reg_res: 1 sub-model
# svm_res: 5 sub-models
# Outcome: latency (numeric)as_tibble(tree_frogs_data_st)
```
> as_tibble(tree_frogs_data_st)
# A tibble: 429 x 8
latency lin_reg_res1 rf_reg_res1 svm_res4 svm_res2 svm_res3 svm_res5 svm_res1
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 22 36.0 49.9 71.5 69.1 40.9 71.5 71.5
2 360 165. 152. 72.2 81.7 121. 73.1 72.2
3 106 122. 117. 72.2 75.9 99.2 72.5 72.2
4 180 145. 134. 70.7 81.6 117. 72.0 70.8
5 39 85.8 74.5 72.2 71.8 72.7 72.0 72.2
6 214 127. 111. 70.7 74.3 99.1 71.2 70.7
7 50 31.0 47.3 70.7 67.8 38.0 70.5 70.7
8 224 121. 111. 70.7 74.0 95.6 71.1 70.7
9 63 36.9 49.2 71.5 69.2 41.3 71.5 71.5
10 33 34.0 48.5 72.2 69.4 40.2 71.8 72.2
# ... with 419 more rowsこれが実際のデータでTibble形式です。
3.3 学習
学習していきます。
tree_frogs_model_st <- tree_frogs_data_st %>% blend_predictions() tree_frogs_model_st
> tree_frogs_model_st
-- A stacked ensemble model -------------------------------------
Out of 7 possible candidate members, the ensemble retained 2.
Lasso penalty: 0.1.
The 2 highest weighted members are:
# A tibble: 2 x 3
member type weight
<chr> <chr> <dbl>
1 svm_res3 svm_rbf 0.860
2 rf_reg_res1 rand_forest 0.400
Members have not yet been fitted with `fit_members()`.tree_frogs_model_st <- tree_frogs_model_st %>% fit_members()
これで、できたオブジェクトを使って新データでの推測ができるようになりました。
tree_frogs_test <- tree_frogs_test %>% bind_cols(predict(tree_frogs_model_st, .))
結果をグラフで表示してみます。
ggplot(tree_frogs_test) +
aes(x = latency,
y = .pred) +
geom_point() +
coord_obs_pred()
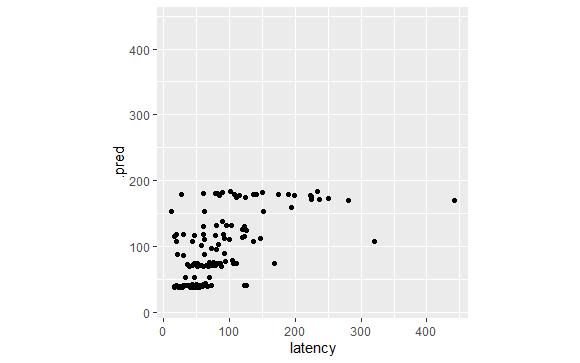
個々のモデルよりスタックした方がよくなったのでしょうか?
member_preds <-
tree_frogs_test %>%
select(latency) %>%
bind_cols(predict(tree_frogs_model_st, tree_frogs_test, members = TRUE))
member_preds %>%
pivot_longer(-latency, names_to = "name", values_to = "val") %>%
ggplot() +
aes(x = latency,
y = val,
col = name) +
geom_point() +
coord_obs_pred()
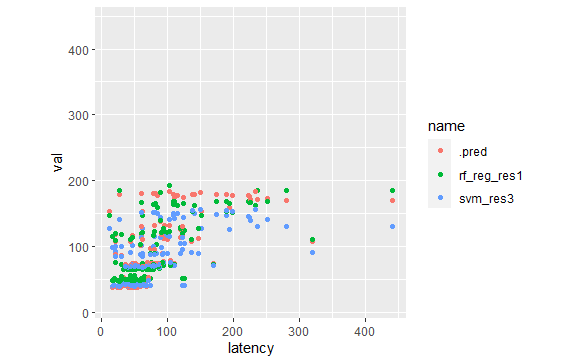
??果たして、個別モデルよりスタックした方がよくなったと言えるのでしょうか?よくわかりません。。。
4. さいごに
最後は、よくわかりませんでしたが、アンサンブルでモデルが良くなることはあると思いますので、これからもう少し勉強してみます。今回は、ある程度使い方を知ったということで。。。

