【R】Rで機械学習 mlrの紹介
2020年5月24日
1.はじめに
Hefin Rhys氏によるRの紹介YouTubeから、Introduction to the mlr package in Rを見てみました。
mlrは機械学習のパッケージで非常に多機能。インストールにもたくさんの時間がかかります。
Youtubeのほぼ写しです。
2.データ
データは、mclustパッケージに含まれるdiabetesという糖尿病に関するデータです。糖尿病については詳しく知りませんが、通常、臨床的糖尿病、化学的糖尿病のタイプに対して、グルコース、インスリン、sspg(steady state plasma glucose)が変数としてあります。この3つの変数から、どのタイプかを予測するのがこの課題です。
library(mlr) library(tidyverse) # Load data data(diabetes, package="mclust") diabetesTib <- as_tibble(diabetes) diabetesTib summary(diabetesTib)
Tibbleにしたデータ
> diabetesTib
# A tibble: 145 x 4
class glucose insulin sspg
<fct> <dbl> <dbl> <dbl>
1 Normal 80 356 124
2 Normal 97 289 117
3 Normal 105 319 143
4 Normal 90 356 199
5 Normal 90 323 240
6 Normal 86 381 157
7 Normal 100 350 221
8 Normal 85 301 186
9 Normal 97 379 142
10 Normal 97 296 131
# ... with 135 more rows> summary(diabetesTib)
class glucose insulin sspg
Chemical:36 Min. : 70 Min. : 45.0 Min. : 10.0
Normal :76 1st Qu.: 90 1st Qu.: 352.0 1st Qu.:118.0
Overt :33 Median : 97 Median : 403.0 Median :156.0
Mean :122 Mean : 540.8 Mean :186.1
3rd Qu.:112 3rd Qu.: 558.0 3rd Qu.:221.0
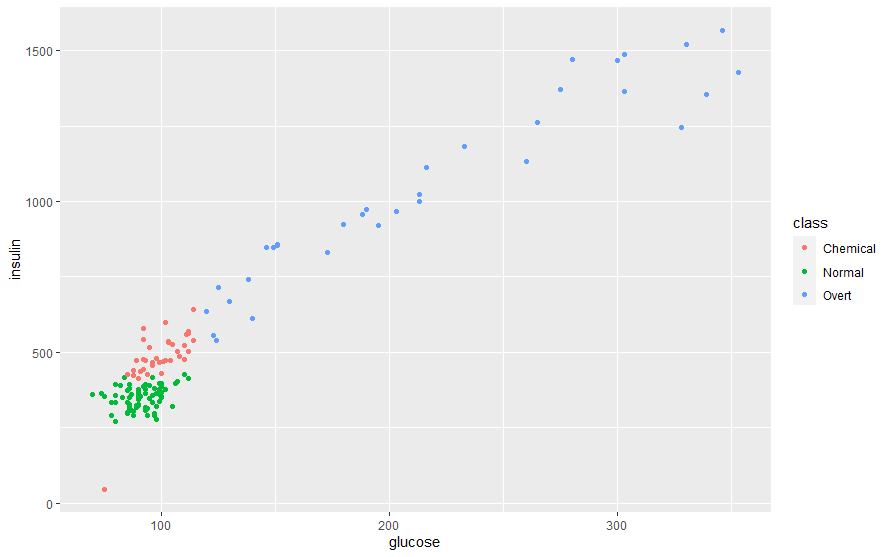
Max. :353 Max. :1568.0 Max. :748.0 データをプロットして確認してみます。
#Plot data ggplot(diabetesTib, aes(glucose, insulin, col = class)) + geom_point()
ggplot(diabetesTib, aes(sspg, insulin, col = class)) + geom_point()

ggplot(diabetesTib, aes(sspg, glucose, col = class)) + geom_point()

3.学習
まず、タスクを設定します。
#Define a tast diabetesTask <- makeClassifTask(data = diabetesTib, target = "class") diabetesTask
> diabetesTask
Supervised task: diabetesTib
Type: classif
Target: class
Observations: 145
Features:
numerics factors ordered functionals
3 0 0 0
Missings: FALSE
Has weights: FALSE
Has blocking: FALSE
Has coordinates: FALSE
Classes: 3
Chemical Normal Overt
36 76 33
Positive class: NA学習してみます。
#Define a learner
knn <- makeLearner("classif.knn", par.vals = list("k" = 2))
#Lsit MLR's algorithms
listLearners("classif")$class
#Training model
knnModel <- train(knn, diabetesTask)
#Testig performance (Bad way)
knnPred <- predict(knnModel, newdata = diabetesTib)
結果のパフォーマンスを見てみます。
performance(knnPred)
> performance(knnPred)
mmce
0.0689655293%程度の正解です。上にBadWayと書いてあるように、トレーニングのデータとテストのデータを同一にしているので、パフォーマンスが良いのは当然です。そこで、もう一度データをサンプリングしなおして学習してみます。
# CrossVaridation
kFold <- makeResampleDesc("RepCV", folds = 18, reps = 58)
kFoldCV <- resample(learner = knn, task = diabetesTask,
resampling = kFold)
calculateConfusionMatrix(kFoldCV$pred)
> calculateConfusionMatrix(kFoldCV$pred)
predicted
true Chemical Normal Overt -err.-
Chemical 1686 206 196 402
Normal 168 4240 0 168
Overt 316 0 1598 316
-err.- 484 206 196 886これぐらいが妥当なのでしょうか?精度を向上させるためには、ここからチューニング等をすべきですが、この動画では紹介としてここまででした。
